近日,复旦大学与四川大学华西医院团队合作在国际权威期刊《科学》杂志上发表了一项突破性研究。这项研究将深度学习技术与冷冻电镜(Cryo-EM)技术结合,通过筛选具有折叠潜力的RNA序列,成功解析了天然RNA多态性组装的分子机制。这一成果不仅初步解决了长期困扰RNA结构研究的技术瓶颈,也为RNA生物学研究开辟了新的方向。复旦大学智能复杂体系基础理论与关键技术实验室博士生吴浩、许晟为共同第一作者,孙思琦为共同通讯作者。

RNA结构研究的挑战与机遇
RNA作为生命活动的核心分子,其复杂的三维结构与动态多态性直接调控基因表达、病毒复制等关键过程。然而,传统RNA结构解析面临两大难题:
1. 实验成本高昂:冷冻电镜虽能解析高分辨率结构,但需耗费大量资源筛选可形成稳定构象的RNA序列。
2. 预测精度不足:现有算法依赖保守二级结构(如Rfam数据库),但同一家族内仅少数序列能形成刚性三级结构。
在此背景下,团队研发了CRAFTS模型(Contrastive RNA learning For sTructure Screening)。通过深度学习技术,该模型能够从海量RNA序列中挖掘潜在的折叠规律,为冷冻电镜实验提供精准的筛选工具,从而显著提高效率并降低实验成本。
CRAFTS模型:技术突破与创新亮点
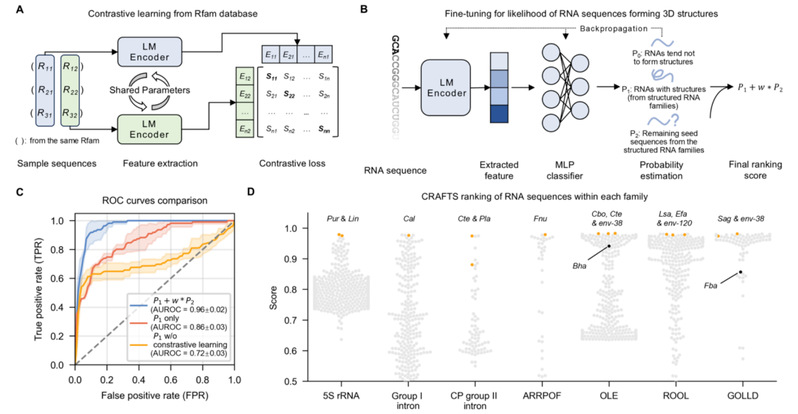
图2 CRAFTS的性能评估,分析RNA家族结构特征。A. 对比学习用于确定输入对是否属于同一RNA家族。B. 微调模型以从每个RNA家族的序列中提取结构特征。C. 基准数据性能表现:平均ROC曲线及标准差(基于5折交叉验证)。D. 对5S rRNA、I类内含子、CP II类内含子等家族分析。通过实验结构测定验证的序列以橙色标注。
CRAFTS模型在技术架构和创新应用上展现了显著的突破性进展。模型基于超过10亿条非冗余RNA序列,这些数据来源于RNAcentral、NCBI等权威数据库,通过自监督学习提取RNA序列的语义特征,生成深度表征。在训练策略中,CRAFTS模型采用了对比学习的方法,通过构建超过900万对RNA序列,最大化同一RNA家族内序列的相似性,同时最小化不同RNA家族序列之间的相似性,从而精准提取家族特异的结构特征(见图2A)。此外,模型利用Rfam数据库中4,038个RNA家族的数据进行训练,并整合了未解析的RNA种子序列作为伪负类,显著扩展了训练数据的范围(图2B)。这种数据增强策略大幅提升了模型的泛化能力,尤其是在数据稀缺的场景下,模型表现尤为突出。例如,在仅有364个正样本的训练集中,CRAFTS通过5折交叉验证展现了高鲁棒性,测试结果的标准差低于0.03(见图2C)。

图3 冷冻电镜结构。A.ARRPOF 二聚体双构象冷冻电镜结构。B.OLE 二聚体冷冻电镜结构。C.ROOL 六聚体和八聚体冷冻电镜结构。D.GOLLD 十二聚体冷冻电镜结构。
在实际应用中,CRAFTS模型不仅在经典RNA家族(如5S rRNA、I型内含子和II型内含子)中表现优异,还成功应用于ARRPOF、OLE、ROOL和GOLLD等新RNA家族的结构筛选(见图2D)。研究团队通过冷冻电镜技术解析了这些RNA家族的结构,发现ARRPOF和OLE可以形成二聚体,而ROOL和GOLLD则分别能够形成六聚体、八聚体和十二聚体的纳米结构(图3)。
总结与展望
CRAFTS模型与冷冻电镜技术的深度协同,成功突破了RNA结构筛选中的“盲盒”困境,为RNA结构生物学研究提供了全新的工具。这一研究成果不仅提升了RNA三级结构解析的效率,还为探索RNA多态性在生命活动中的潜在功能开辟了新的可能性。
尽管研究取得了重要进展,但仍面临一定局限性。研究表明,CRAFTS能够有效提取ARRPOF RNA家族中与三级结构形成相关的特征(如P4和P6的变异,以及P10额外核苷酸可能导致的空间位阻和结构破坏),但在OLE、ROOL和GOLLD等RNA家族中,序列一致性变异的相关性尚不明确。这表明CRAFTS模型对RNA家族间的广泛泛化能力仍有待提升。其主要限制因素在于目前蛋白质数据库(PDB)中RNA结构数据的稀缺性。这种数据不足制约了模型的训练和验证,未来需要通过进一步优化模型和增加高质量数据来提升其在不同RNA家族中的预测能力。
论文标题:Cryo-EM reveals mechanisms of natural RNA multivalency
论文链接:https://www.science.org/doi/10.1126/science.adv3451











