近年来,生物节律被证明与人类的健康息息相关,受到了越来越多科研人员的重视,2017年诺贝尔生理学或医学奖还授予了生物节律开创性的工作【1】。除了人们熟知的昼夜节律(周期约24小时),生物体内所展现的其他有节奏的规律性变化都可以被统称为生物节律,例如:神经元的动作电位、基因-蛋白转录翻译过程、维持生命的心跳等。生物节律的频率和振幅作为两个重要的物理量分别决定了上游信号的特征和强度,并进一步影响下游活动【2】。因此,由于各种原因所导致的不正常的频率或振幅会造成节律紊乱进而引起各种生理疾病,如:睡眠障碍、二型糖尿病等【3】。所以,如何精确调控生物节律的频率和振幅使其恢复正常成为了一个重要的科学问题。早期,就有学者通过大规模计算,开创性地研究了频率和振幅可调性与节律系统反馈回路模式之间的联系【4】;近期,也有学者提出了一些生物学上可行的频率振幅控制器,通过计算也给出了验证【5】。但是,有关生物节律频率和振幅的调控尚缺少系统性、普适性的理论进展,更为重要的是,仍然缺少一种基于数学理论的计算方法用于提前设计能够在复杂时空系统精确调节频率振幅的控制器。
针对这些问题,2021年10月8日,复旦数学的秦伯韡博士、赵磊博士和林伟教授于《自然・通讯》Nature Communications 在线发表了研究论文(Research Article)“生物振荡系统的调频调幅控制器及最优能量实现” A frequency-amplitude coordinator and its optimal energy consumption for biological oscillators。该成果用严格数学理论和普适计算方法准确地设计了可用于调节时空生物振荡器内在频率和振幅的控制器。该方法可以普适性地应用于不同尺度带有扩散现象的生物节律模型,实现频率振幅的可算可控。相关成果也将有关生物振荡系统频率振幅调制的早期理论成果【6】成功推至“时空复杂系统”之中。
研究人员针对基因调控网络抽象时空模型,利用米氏调节(Michaelis-Menten regulation)实现了对蛋白表达水平的调控。在该模型中,一旦蛋白浓度超过某一阈值便会进入“癌症区”。利用精准设计的控制器可以使蛋白浓度缓慢降低至正常水平(图1)。

图 1 抽象基因-蛋白互作时空模型中的振幅调控。
随后,研究人员运用动力系统理论阐明了为何这样的控制方法可以有效调节频率和振幅,同时给出了详细的计算方法。对于所研究的由分岔所产生的节律振荡,研究人员利用了米氏函数的线性区域对生物振荡器的内蕴线性作用进行了扰动,这样的扰动可以对频率和振幅产生较为可观的变化。而运用中心流形、规范型理论,能够通过严格的计算过程揭示扰动强度和频率振幅变化量之间的关系(图2)。

图 2 通过动力学理论实现频率和振幅信息的提取。
运用这类方法,研究人员也实现了对时空神经元模型(FitzHugh-Nagumo)的频率调节,验证了该方法的普适性(图3)。之后,进一步揭示了控制器所遵循的一般规律:频率-振幅调节器是对内蕴线性作用的一种扰动,而这样的扰动只会同时加强或减弱内蕴作用(图4)。因此,所设计的控制器可以被视为是一种放大器或阻尼器。最后,研究人员还考虑了控制器的能量消耗,并能够以此为依据挑选出最优的控制器。
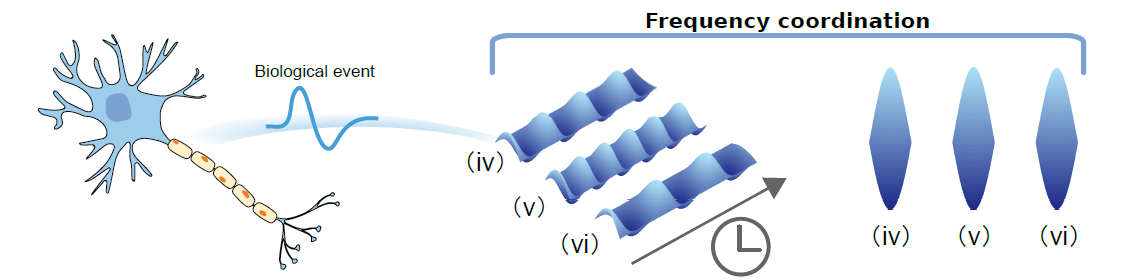
图 3 神经元模型中的频率调节。
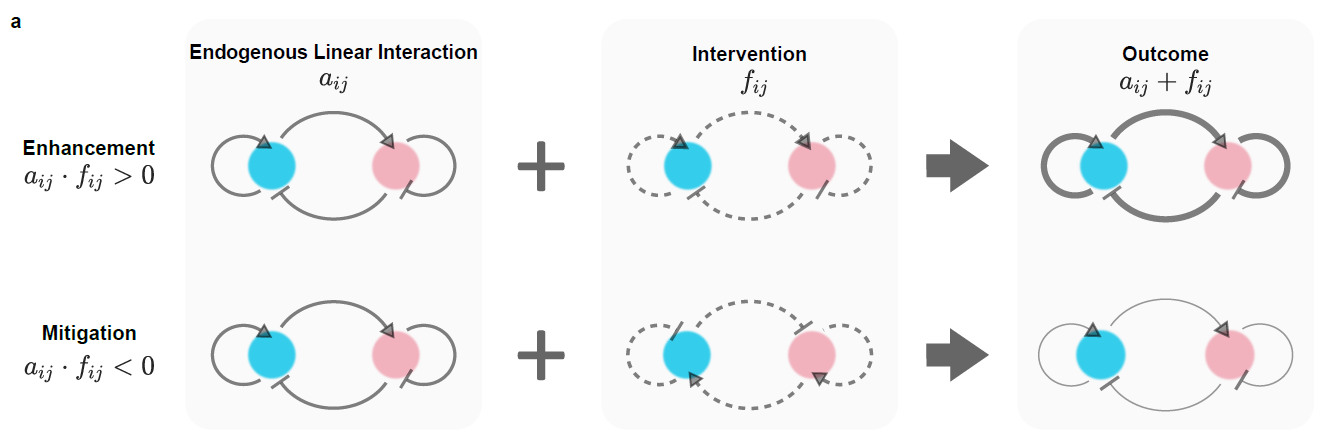
图 4研究所设计控制器的一般规律。
该工作中,研究人员将生物科学问题与应用数学方法紧密结合,提出了一种可行的调控手段,可以为生物节律调频调幅的实验性研究提供理论支撑,有望加速这一方向的后续发展。复旦数学的博士后秦伯韡博士是该工作的第一作者,秦伯韡博士、林伟教授是该工作的共同通讯作者。本研究得到了国家自然科学基金委、教育部脑科学前沿中心、科技部重点研发项目、上海市市级重大专项、中国博士后基金委、教育部非线性数学模型与方法重点实验室、教育部计算神经科学与类脑智能重点实验室等的支持。
原文链接:https://www.nature.com/articles/s41467-021-26182-2
参考文献:
Callaway, E. & Ledford, H. Medicine Nobel awarded for work on circadian clocks. Nature550, 18 (2017).
Hansan, A. S. & O’Shea, E. K. Limits on information transduction through amplitude and frequency regulation of transcription factor activity. eLife4, e06559 (2015).
Bass, J. & Takahashi, J. S. Circadian integration of metabolism and energetics. Science330, 1349-1354 (2010).
Tsai, T. Y.-C. et al. Robust, tunable biological oscillations from interlinked positive and negative feedback loops. Science321, 126-129 (2008).
Tomazou, M.et al. Computational re-design of synthetic genetic oscillators for independent amplitude and frequency modulation. Cell Systspans6, 508-520.e5 (2018).
Ge, T. et al. Achieving modulated oscillations by feedback control. Physical Review E90, 022909 (2014).
